前言
精读一下文章, 记录一些东西,其实好像就是翻译一下。
今天的主角是这篇Single-cell full-length total RNA sequencing uncovers dynamics of recursive splicing and enhancer RNAs,发表在Nature Communication杂志上,是日本理化研究所(RIEKN)的成果。
这篇文章主要介绍了他们建立的RamDA-seq测序方法,号称是第一个针对单细胞的全长总RNA组测序方法。
主要亮点
- RamDA-seq 对于non-poly(A) RNA的sensitivity较高,并且得到的reads能够更完整地覆盖transcript(near-complete full-length transcript coverage)
- RamDA-seq 能够找到 >300kb的内含子的recursive splicing事件和增强子RNA,以及它们在单细胞中特异性的作用模式。
背景
生物体中存在的总RNA丰度极高并且参与各种生命过程的调控。自然能够越详细地得到这些数据越能帮助人类理解生命过程,尤其是单细胞水平上。
目前当然也有很多单细胞针对RNA的测序方法,但是绝大多数都是通过捕获polyA测得转录谱,对于调控性质的enhancerRNA (eRNA) 和long non-codingRNA (lncRNA) 就捕获得很少。这怎么能行呢(:зゝ∠)这还怎么研究调控了。
目前市面上已有报道的SUPer-Seq倒是说它们用连接了随机引物的oligo-dT钓到了一些non-polyA RNA, 比如circRNA什么的,那个sensitivity都太低,没眼看。当然,怎么去除rRNA也是一个大问题,毕竟这些占据了总RNA的大部分,但它们携带的信息又没太多价值。
作者说为了解决这个问题并且还能获得比较高的sensitivity,他们使用了一个新方法叫reverse transcription with random displacement amplification (RT-RamDA, 随机替代逆转录扩增),并且用的引物是not-so-random primers (NSRs, 不辣么随机引物)。据说这个不辣么随机引物可以在随机启示扩增的同时减少来自rRNA的cDNA,引用两篇文献1、2,嗯懒得看,猜测是用计算方法筛选了一下随机引物,把更有可能匹配上rRNA的那些短引物(6bp)都去掉了。
后来看了一下,对就是用计算方法把map上rRNA的短引物都去除了
结果部分
那么问题来了,什么是RT-RamDA
先上图

原理从图上看还是比较清晰的,由于NSR引物的存在,poly-A / non-polyA RNA都能被钓到,而rRNA由于既缺乏polyA尾,NSR也捕获不到。因此这个方法能够较为准确地富集transcripts 和一些较长的RNA片段。之后就是常见的后随链合成、纯化和建库步骤,此处不表。
既然是新方法,避免不了先和已有方法比较一下,做一些benchmark。
Benchmark
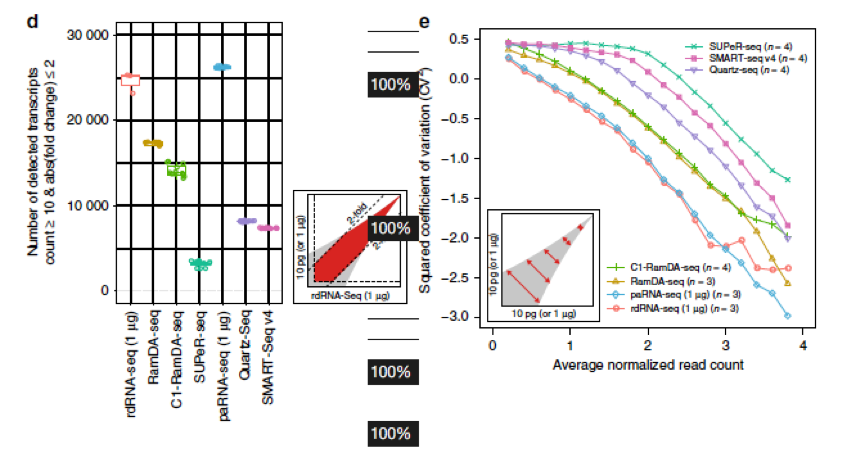
作者用上述流程建了一个库,大概包含有10pg的mESC总RNA,然后比较他们的RamDA-seq和C1-RamDA-seq与已有方法的一些指标的区别。
已有方法作者选择了上文里提到的SUPeR-seq,以及基于oligo-dT的方法如SMART-seq v4, Quartz-seq。并且作者用了1μl总RNA做了去除rRNA的总RNA bulk 测序(rdRNA-seq)和富集polyA的RNA bulk测序(paRNA-seq)作为分析的上限(可以理解成阳性对照吧)。
选取的比较指标主要有:
- 找到的transcript数量,transcript加了限定条件必须是和rdRNA-seq找到的相比表达量变化在两倍范围内的
- 变异系数平方(CV^2)和表达量的关系,用来检测可重复性(reproductivity)?
- 和rdRNA-seq的表达量correlation
- 其他。。spike-in什么的
结论是相比其他单细胞方法来说和bulk总rna测序结果最为接近,鲁棒性和sensitivity都优于现有方法。
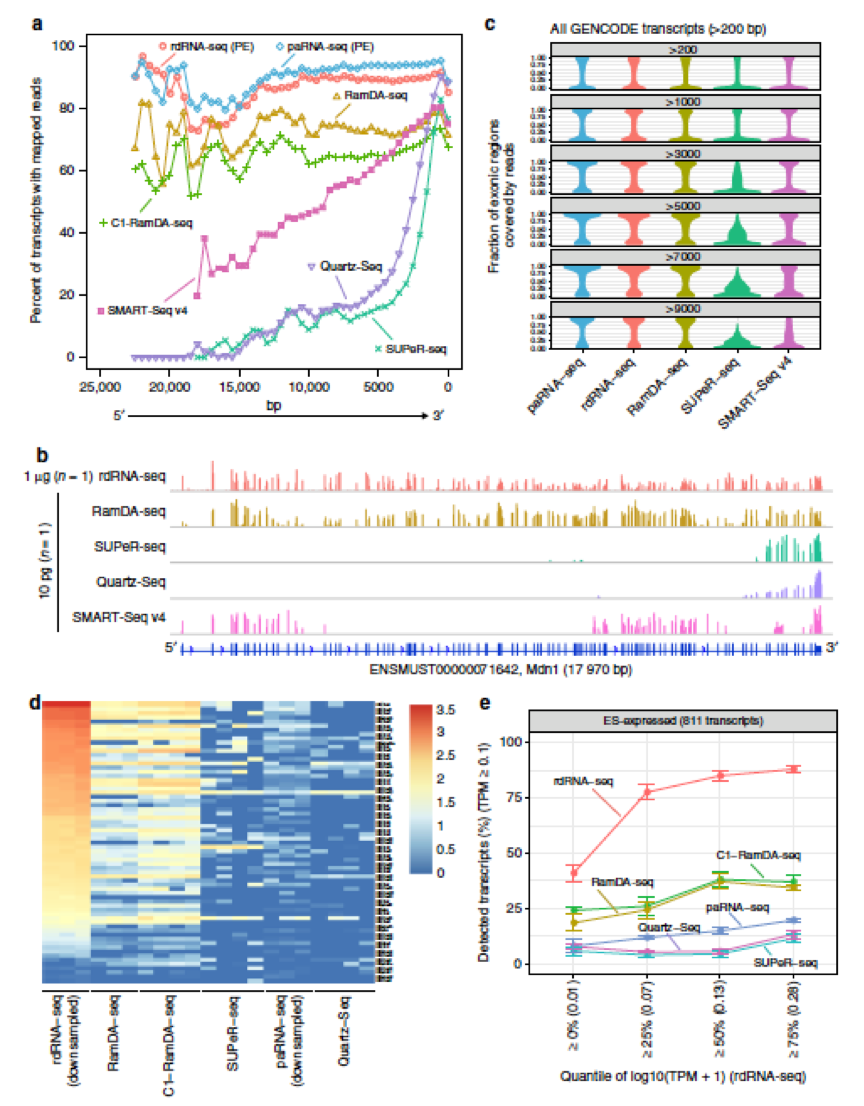
当然,既然号称是能提到更多transcripts和non-polyA RNA的方法,reads coverage的对比也不可少
fig2里5‘-3‘的reads coverage可以很明显地看到基于oligo-dT的几个方法都有3’bias,而新方法更接近bulk测序结果。
对较长transcripts外显子区域覆盖率相对SUPeR和SMART-Seq都更高。
生物学问题 - 细胞分化中的应用
Benchmark做完,说明这个好用,仅仅到此为止只是一个普通的方法学文章。当然还要和生物学意义结合起来。
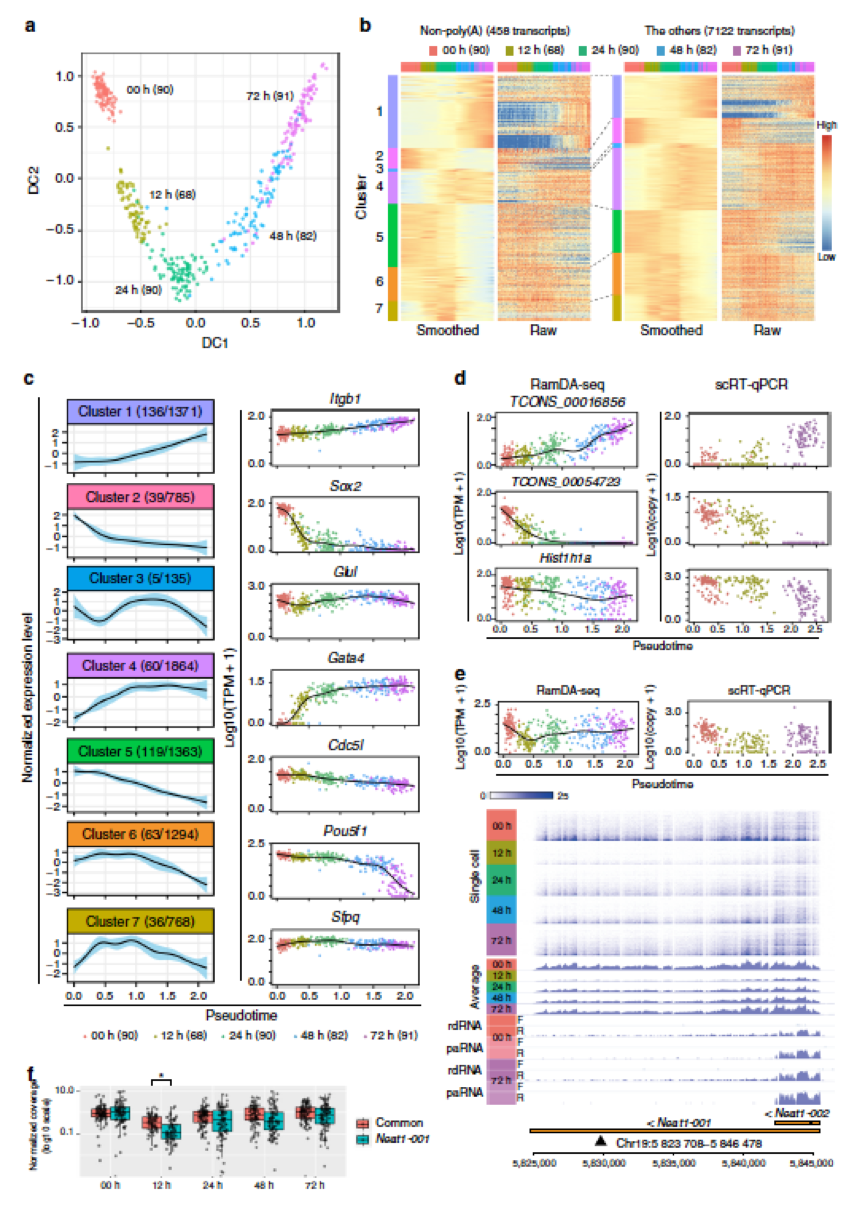
作者用多时间点取样的mESC做了一批单细胞数据。这种数据的好处应该是可以跟踪这批细胞在不同时间点的分化状态。分化刚好是一个transcripts调控变动比较剧烈的过程,用这个新方法可以说是比较合适。
首先是一个diffusion map。可以看到各个时间点的聚类结果可以说是很漂亮了,但是没仔细看方法,不太确定其中的人为因素。他们这套数据一个亮点就是还存在比较丰富的non-polyA RNA 表达,并且确实在各个阶段都检查到了,在观察调控上可以说很有优势。
并且他们还做了scRT-QPCR来验证其中一部分的non-polyA RNA表达量,其中还包括了两条未注释过的RNA。作者放话说下一步就是要利用这批数据做一个生物功能富集出来。
不过鉴于这篇文章还是偏方法学,所以作者在这里找了两条应该是已有研究比较充分的一条长的transcripts- Neat1-001 它有一条较短的isoform Neat1-002 。从结果上看似乎比较符合作者的预期,但是我不太了解这个基因,所以作者说啥就是啥吧(:зゝ∠)
生物学问题 - 递归剪切 Recursive splicing
介绍完细胞水平上的应用,接下来就是新方法得到的数据在更细微尺度上的应用。
作者选择了RS,也即是脚注里介绍的recursive splicing的检测。
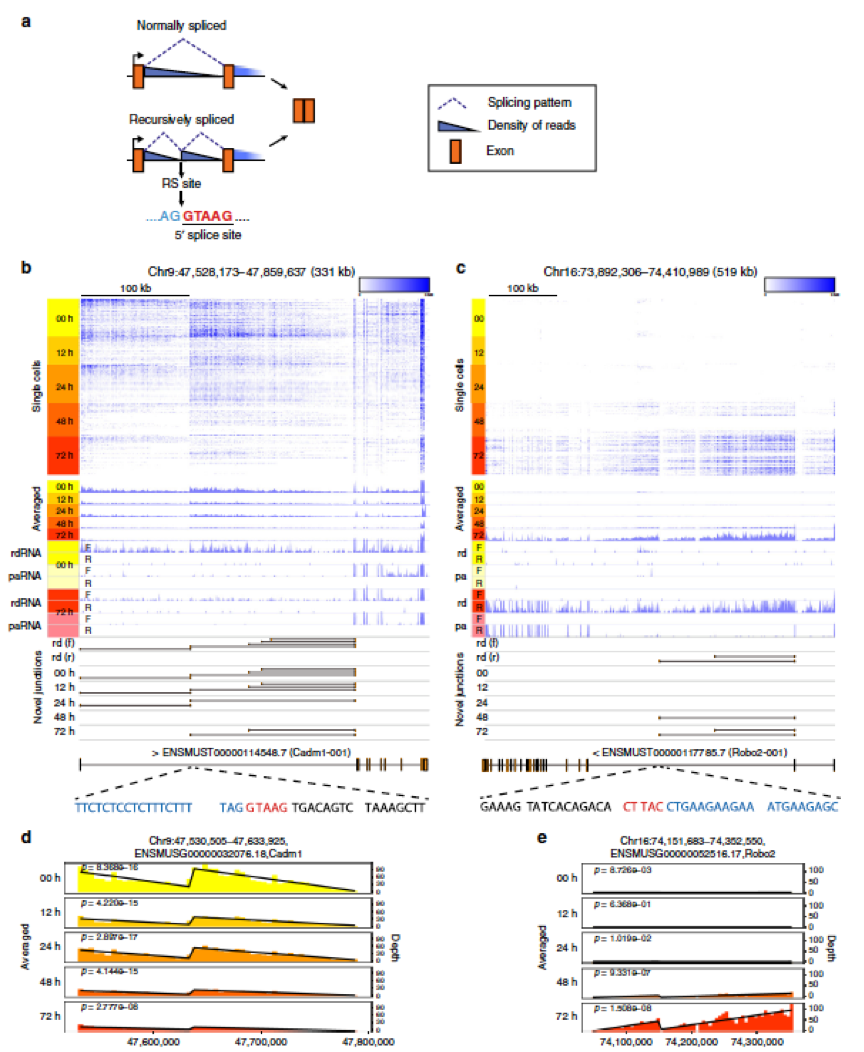
他们假设如果这个现象能被他们的方法观测到,那么reads coverage应该是呈现锯齿状。
作者通过线性回归模型拟合锯齿(有点骚)应该是找到了一些基因,然后高亮了这三个,Cadm1、Robo2和 Magi1 ,说这三个都是有文献报道和最初在mESCs里做出来的有RS现象的基因。图上只展示了 Cadm1 和 Robo2 这两个基因。
可以看到两个基因的bulk结果都有锯齿pattern,但在时间点上bulk数据不太明显。但是从单细胞数据上看却能明显地看到它们在不同的时间段的锯齿情况。
生物学问题 - 增强子RNA Enhancer RNAs
non-polyA RNA里的一部分premature RNA已经在上一个段落里观测过了,现在来看看另一个部分,enhancer RNA。

ES-active enhancer 逐步降低,ES-inactive 一直保持低水平表达。在这个数据集里和预期一致。并且,fig5b里也能看到background也即是随机的基因组区域一直都是低水平。
结合前人在eRNA激活位点富集的条件特异激活的转录因子的DNA结合motif的研究,作者也尝试搜索了一下ESC中激活的eRNA和motif, 并且和rdRNA-seq结果进行比较。召回率还是比较可观的,75%左右吧。发现相当一部分在self-renewal和多能性(pluripotency)相关的TF上。
后面还是照例做了时间序列研究,热图和右边的曲线图能看到还是找到了一些东西。作者说了几个例子表示这个结果还比较可信,但是具体有多可信就。。
Discussion
主要看看作者介绍的一些局限性吧。
- 这个方法不能改造成UMI-based方法。
- 和其他单细胞测序方法比起来测序深度需要更深一些
- rRNA还是会有,大概在10-35%这个量级
具体测多深呢,作者说在subsampling simulation中,
- > 1M reads per cell 就能找到比其他方法更多的transcripts
- ~ 4M reads per cell 就能找到上面介绍的各种包括新non-polyA transcripts啦,RS啦各种东西
作者说鉴于4M就是个96孔板一个run测序普通程度,所以还行吧!
然后吹了一波还可以做什么什么,基本上关于RNA的都说了一遍,都能看
作者提到了似乎对于同一个长转录本,细胞和细胞的剪切方式可能是不一样的,有的用RS方式,有的用normal方式(Supplementary Fig17)。嗯然后说用线性回归找锯齿确实还是有点粗糙。。。
(可能前面说的细胞异质性其实只是线性回归找的不准确?毕竟靠计算方法找感觉还是有一点点不靠谱,两个模型有时候差距很小。。)
基本情况就酱~
Footnotes
Recursive splicing
根据这篇文章的定义:
Recent work in human and fruitfly tissues revealed that long introns are extensively processed cotranscriptionally and in a stepwise manner, before their two flanking exons are spliced together. This process, called “recursive splicing,” often involves non-canonical splicing elements positioned deep within introns, and different mechanisms for its deployment have been proposed.
Recursive splicing,直译递归剪切,指的是一种在果蝇和人类中存在的针对长内含子的剪切方式。与短内含子直接剪除不同,长内含子是一段一段“递归地”被剪除的。
Diffusion map
Introduction for diffusion map of Wikipedia
如有问题/建议/意见,欢迎留言交流
版权声明: 本博客所有文章除特别声明外,均采用CC BY-NC-SA 3.0 CN许可协议。转载请注明出处!